DNA结构是什么?关于DNA结构的科普介绍

DNA(DeoxyriboNucleic Acid)指脱氧核糖核酸,由两条平行的链组成,两条链互相绕成螺旋状。双螺旋结构中磷酸与脱氧核糖通过磷酸二酯键链接交替排列在结构外侧,构成DNA分子的基本骨架,内侧碱基通过氢键相连形成碱基对,碱基对形成遵循碱基互补配对原则。DNA是由四种脱氧核糖核苷酸组成而成的大分子聚合物,是主要的遗传物质。

DNA的一级结构
一级结构是指构成核酸的四种基本组成单位——脱氧核糖核苷酸,通过3',5'-磷酸二酯键彼此连接起来的线形多聚体。
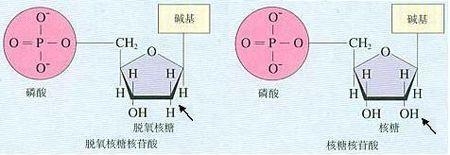

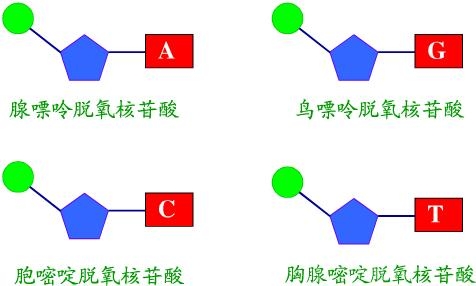
DNA的二级结构
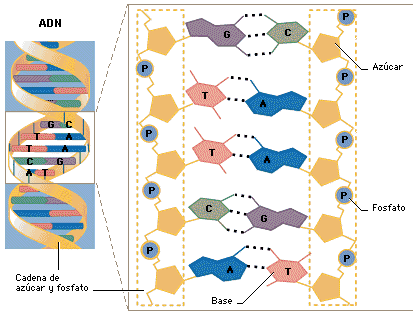
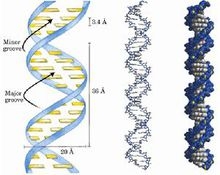
碱A与T之间可以形成两个氢键,G 与C之间可以形成三个氢键,使两条多聚脱氧核苷酸形成互补的双链,由于组成碱基对的两个碱基的分布不在一个平面上,氢键使碱基对沿长轴旋转一定角度,使碱基的形状像螺旋桨叶片的样子,整个DNA分子形成双螺旋缠绕状。碱基对之间的距离0.34nm,10个碱基对转一周,故旋转一周(螺距)3.4nm,这是β-DNA的结构,在生物体内自然生成的 DNA几乎都是以β-DNA结构存在。
单链DNA
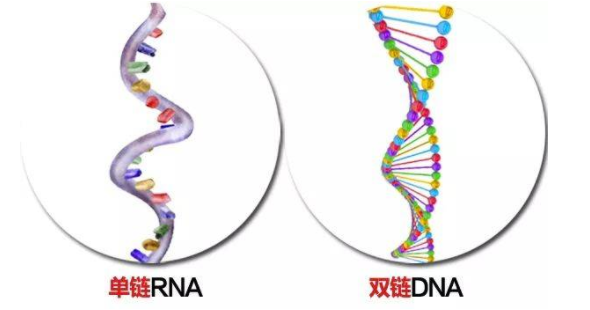
大部分DNA以双螺旋结构存在,但一经热或碱处理就会变为单链状态。单链DNA就是指以这种状态存在的DNA。单链DNA在分子流体力学性质、吸收光谱、碱基反应性质等方面都和双链DNA不同。某些噬菌体粒子内含有单链环状的DNA,这样的噬菌体DNA在细胞内增殖时则形成双链DNA。
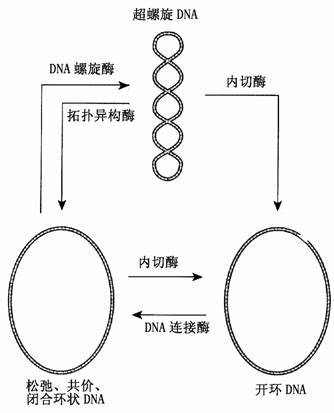
闭环DNA
一个长的双链DNA(双螺旋的)首尾是相接,没有断口,也称作超螺旋DNA。是游离在细胞核外的双链可自我复制的DNA环状物。在基因工程上,称它为质粒用于承载DNA片段,多存在细菌等拟合生物中。
双螺旋结构的特点
1、DNA是反向平行的互补双链结构,它的两条多聚核苷酸链在空间排布呈反向平行,碱基位于内侧,亲水的脱氧核糖基和磷酸基位于外侧,碱基间以A-T和G-C的方式互补配对;
2、DNA双链是右手螺旋结构,DNA的两条多核苷酸链反向平行围绕同一中心轴互相缠绕,呈右手螺旋;
3、疏水力和氢键维系DNA双螺旋的稳定,横向稳定靠碱基间的氢键维系,纵向靠碱基平面间的疏水性堆积力维持。
DNA分子中碱基数量计算的规律
根据碱基互补配对原则引出的关于碱基比率和数量的计算是本节的重点之一,可以根据以下规律解决这一问题。
规律一:互补的两条链之间碱基数量相等,即A=T,G=C。
规律二:任意两个不互补的碱基之和占碱基总数的50%,即A+G=T+C=A+C=T+G=50%。
规律三:两个不互补的碱基之和比值相等,即(A+G)/(T+C)=(A+C)/(T+G)=1。
规律四:一条链中互补碱基的和等于另一条链中这两个碱基的和,即A1=T2,T1=A2或A1+T1=A2+T2。
规律五:一条链中互补的两碱基的和占该单链的比例等于DNA分子双链中这两种碱基的和占碱基总数的比例,
即(G1+C1)/单=I(G+C)/双,(A1+T1)/单=(A+T)/双。
规律六:若一条链中(A1+G1)/(T1+C1)=K,则另一条链中(A2+G2)/(T+C2)=1/K。
DNA的三级结构
DNA的拓扑结构
这也是DNA存在的一种形式。DNA的拓扑结构是指在DNA双螺旋的基础上,进一步扭曲所形成的特定空间结构。超螺旋结构是拓扑结构的主要形式,可以分为正超螺旋和负超螺旋两类,在相应条件下,它们可以相互转变。
DNA结构的特点
①多样性:DNA分子碱基对的排列顺序千变万化。
一个由n个碱基对组成的DNA分子可能的排列方式有4种。一个最短的DNA分子也有4000个碱基对,可能的排列方式就有4000种。
②特异性:特定的DNA分子具有特定的碱基排列顺序。
不同的生物,碱基对的数目可能不同,碱基对的排列顺序肯定不同。
③稳定性
DNA分子的多样性由DNA分子双螺旋结构中碱基对的排列顺序和数量决定、稳定性是由DNA分子的碱基互补配对以及双螺旋等特点决定的、特异性是由DNA分子的碱基对排列顺序
内含子与外显子
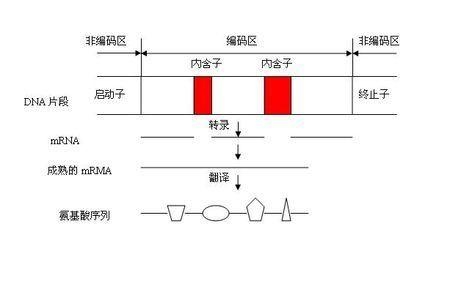
外显子(expressed region),是真核生物基因的一部分,它在剪接后仍会被保存下来,并可在蛋白质生物合成过程中被表达为蛋白质。外显子是最后出现在成熟RNA中的基因序列,又称表达序列。既存在于最初的转录产物中,也存在于成熟的RNA分子中的核苷酸序列。通过确定在多种生物中出现的片段来鉴定编码区域,而外显子的保守性可以作为这种鉴定的基础。
DNA的复制
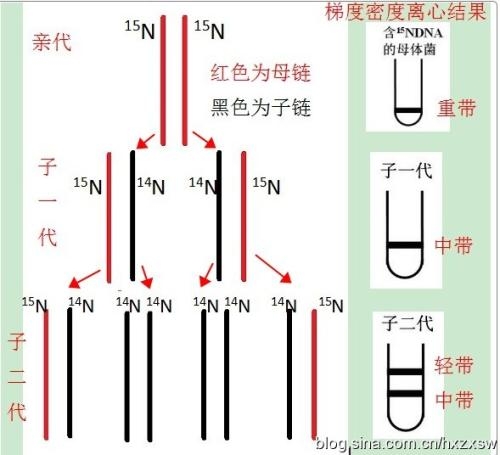
DNA在复制时,需在特定的位点起始,这是一些具有特定核苷酸排列顺序的片段,即复制起始点(复制子)。在原核生物中,复制起始点通常为一个,而在真核生物中则为多个。
DNA复制需要引物(primer):DNA聚合酶必须以一段具有3'端自由羟基(3'-OH)的RNA作为引物,才能开始聚合子代DNA链。RNA引物的大小,在原核生物中通常为50~100个核苷酸,而在真核生物中约为10个核苷酸。
双向复制:DNA复制时,以复制起始点为中心,向两个方向进行复制。但在低等生物中,也可进行单向复制。
半不连续复制:由于DNA聚合酶只能以5'→3'方向聚合子代DNA链,因此两条亲代DNA链作为模板聚合子代DNA链时的方式是不同的。以3'→5'方向的亲代DNA链作模板的子代链在聚合时基本上是连续进行的,这一条链被称为领头链(leading strand)。而以5'→3'方向的亲代DNA链为模板的子代链在聚合时则是不连续的,这条链被称为随从链(lagging strand)。DNA在复制时,由随从链所形成的一些子代DNA短链称为冈崎片段(Okazaki fragment).冈崎片段的大小,在原核生物中约为1000~2000个核苷酸,而在真核生物中约为100个核苷酸。
DNA复制完整过程:DNA解旋酶在局部展开双螺旋结构的DNA分子为单链,引物酶辨认起始位点,以解开的一段DNA为模板,按照5'到3'方向合成RNA短链,形成RNA引物。在引物提供了3'-OH末端的基础上,DNA聚合酶催化DNA的两条链同时进行复制过程,当DNA合成一定长度后,DNA聚合酶水解RNA引物,补填缺口。DNA连接酶将DNA片段连接起来,形成完整的DNA分子。最后DNA新合成的片段在旋转酶的帮助下重新形成螺旋状。
DNA生物学功能
1、DNA通过精准的复制,将遗传信息传递给下一代;
2、DNA在复制过程中有概率发生突变,为生物进化提供了分子基础;
3、DNA能转录成RNA,进而翻译成蛋白质,通过蛋白质实现生命的结构和功能;
发现历史

之后,沃森和克里克从生物大分子的基本单位出发,运用化学规律发现核苷酸之间可能形成的排列方式,着重考虑对整个大分子结构的稳定性具有决定作用的氢键的形成方式,并开始了模型设计。两人测定各种嘌呤和嘧啶的大小、碱基对的排列、氢键的引力以及DNA分子直径、螺距、键角等结构数据,再与DNA衍射图像一一对比设计模型。他们发现四种碱基中,腺嘌呤(A)-胸腺嘧啶(T)对之间可以形成两个氢键连接,鸟嘌呤(G)-胞嘧啶(C)对之间可以形成三个氢键连接,两种配对均是一个双环和一个单环的组合,直径相差较小,并且从这一发现解释了查伽夫定律。最终他们设计出了DNA分子双螺旋结构的模型。这一结构模型可以描述为:脱氧核糖和磷酸交替排列构成DNA的基本骨架,碱基在内侧,两条长链的碱基通过氢键形成碱基对,并且是A与T配对,G与C配对。由此也可以证实一条链是如何作为模板合成另一条互补碱基顺序的链,并且两条链的方向一定是相反的。
沃森和克里克用运用一周的时间构建了DNA结构模型,测量出两种碱基对和DNA长链上每一种键的旋转角度。同时将金属材料制成的模型与X射线拍摄的衍射照片相比较,发现二者完全相符,进一步分析证实了双螺旋结构模型是正确的。
1953年4月,英国的《自然》杂志刊登了沃森和克里克在英国剑桥大学合作的研究成果:DNA双螺旋结构的分子模型,这一成果被誉为20世纪以来生物学方面最伟大的发现,标志着分子生物学的诞生。DNA分子结构的发现,更好地解释了DNA是遗传物质以及在分子水平上阐明了DNA的复制和控制蛋白质合成的功能。
基因工程
步骤一:基因文库内寻找目的基因;
步骤二:进行PCR基因扩增;
步骤三:选取合适的质粒作为目的基因载体;
步骤四:利用切位点相同的限制性内切酶分别对利用PCR扩增技术扩增过的DNA和质粒进行酶切;
步骤五:利用与限制性内切酶切点相对应的DNA连接酶进行目的基因与质粒的组装;
步骤六:重复以上步骤,在质粒中导入抗性基因以及启动子、终止子;
步骤七:利用显微注射技术导入目的基因;
步骤八:利用抗性基因所抗抗生素,选出目的基因成功导入的细胞;
步骤九:单另培养细胞,检查是否可以产生目的基因对应蛋白,如果可以产生,即为目的基因表达成功,在检测其安全性後,可以批量培养
DNA折纸技术
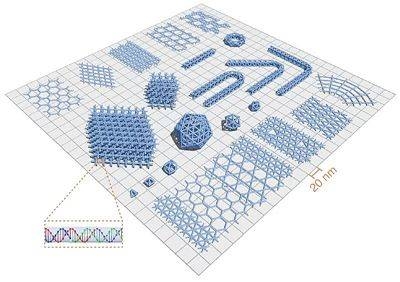
在用DNA制作各种图样时,先借助纳米仪器,绘制折叠物形状,然后用折叠的DNA长链将形状填满。而DNA短链就是固定折叠的DNA长链的”图钉”,有了它们,DNA长链组成的图形才不会散开。用计算机计算作品所需DNA短链的数量后,将这些DNA短链”钉”在长链构成的支架上。然后将DNA长链和短链一起放入一种碱性溶液加热,它们就会自动结合在一起,组合成人们想要的图案。
2006年美国加州理工学院计算机生物工程师Paul Rothemund开发了一种“DNA折纸术”( DNA origami technique),像折叠一条长带子那样,把一条DNA长链反复折叠,形成需要的图形,就像用一根单线条绘制出整幅图画。经过近十年的发展,DNA折纸技术已经变得更加强大了。来自加州理工学院生物工程系的钱璐璐(Lulu Qian)博士早年师从上海交通大学贺林院士,她一直都对生物纳米技术与DNA分子计算感兴趣,几年前就曾与加州理工学院合作,利用人工合成的DNA分子,在试管中制成了当时最复杂的生化电路。其研究组在Science杂志上发表了全新DNA“机器人”的重要成果,解决了现有的机器人不能自动行走等局限问题。来自慕尼黑理工大学的生物物理学家Hendrik Dietz的研究小组则是采用的DNA折纸技术与逐步构建策略,也就是自然界中分子机器采用的方法,例如对称性和多层次的装配过程。这些方法令他的团队“从DNA折纸之前的megadalton单位发展成了gigadalton单位”。
本文转载于:搜狗科学,baike.sogou.com/kexue/d45973792400379656.htm,本内容使用CC-BY-SA 3.0授权,用户转载请注明出处





















